Materials Studio¶
简介¶
Materials Studio(MS)是法国达索BIOVIA品牌下的一款全尺度材料模拟平台。MS拥有优异的操作界面,能够快捷实现模型搭建、参数设定以及结果的可视化分析;MS中融合了多种模拟技术,整合20多个功能模块,实现从电子结构解析到宏观性能预测的全尺度科学模拟研究。历年的更新,MS在功能、效率、精度、使用体验等方面变得更加完善。Perl脚本编写功能使得MS在计算与分析作业变得更加灵活。
安装环境¶
序号 |
集群 |
平台 |
版本 |
位置 |
安装方式 |
|---|---|---|---|---|---|
1 |
hpckapok1 |
cpu |
23.1 |
/share/software/BIOVIA/MaterialsStudio23.1/ |
软件包安装 |
使用方法¶
小技巧
以下为命令行解释,请勿直接复制运行,作业内容请保持与脚本作业目录一致
slurm作业提交¶
1.CASTEP、Dmol3等模块的实现方式¶
1.登陆到Linux机器上进行脚本提交作业;通过调用各个模块的运行命令,设置相应的核数进行计算;比如CASTEP模块为例(Dmol3模块方式一样),我们需要在Windows的客户端MS创建计算需要的输入文件,创建的方式如图所示:
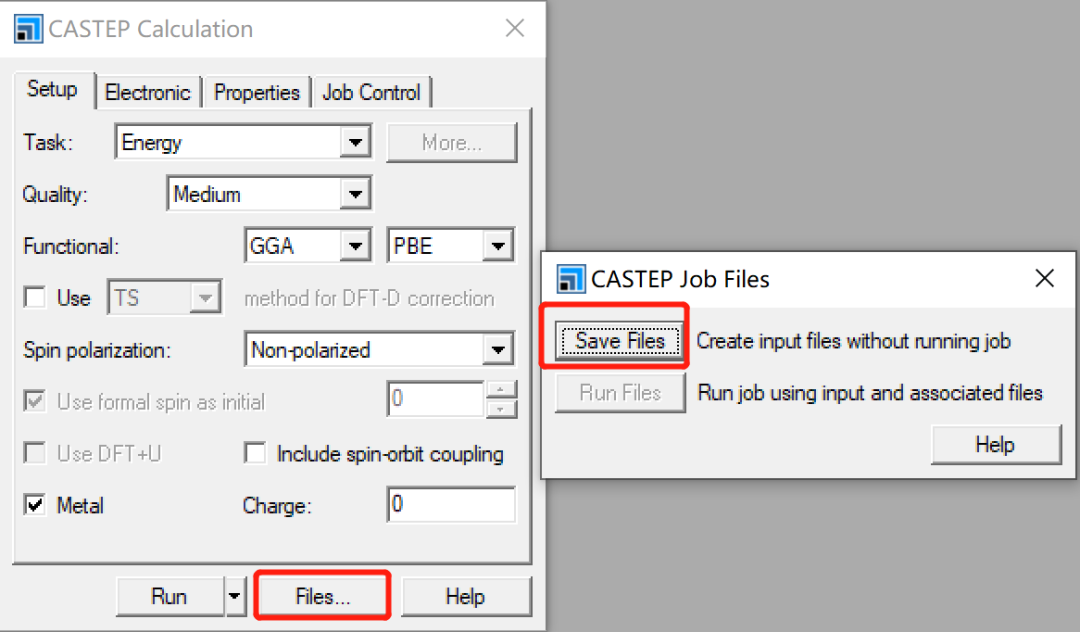
创建完成之后,在MS界面上我们只能看到几个文件,但是很多是隐藏文件,进入到文件夹中,对文件显示方式进行设置,显示隐藏的文件,上传到作业目录
2.创建作业脚本ms.slurm,传输文件目录保持一致
#!/bin/bash
#SBATCH --job-name=test3
#SBATCH --mem-per-cpu=8G
#SBATCH --partition=cpuXeon6458
#SBATCH --nodes=1
#SBATCH -n 64
#SBATCH --output=slurm_out.%j
#SBATCH --error=slurm_error.%j
module swap openmpi4 mvapich2/2.3.7
export LD_LIBRARY_PATH=/share/software/BIOVIA/MaterialsStudio23.1/lib:$LD_LIBRARY_PATH
MS_PATH=/share/software/BIOVIA/MaterialsStudio23.1/etc/CASTEP/bin/ #CASTEP模块
#MS_PATH=/share/software/BIOVIA_stu/MaterialsStudio23.1/etc/CASTEP/bin/ #提交较多作业时可用此版本学习
$MS_PATH/RunCASTEP.sh -np 64 test > output #test为导出文件test.param的文件名的前缀
#MS_PATH=/share/software/BIOVIA/MaterialsStudio23.1/etc/DMol3/bin/ #DMol3模块
#$MS_PATH/RunDMol3.sh -np 64 test > output #test为导出文件test.param的文件名的前缀
使用sbatch ms.slurm提交作业
小技巧
1、关于作业完成后导回本地ms客户端结构不变化问题,超算上计算完后把文件夹下载导入到ms客户端,然后点开那个.xsd文件显示到最前面窗口,然后modules的CASTEP模块的analysis里面有structure有个update就可以更新
2、后续计算可以在第一个计算成功完成后开始,作为连续运行,如此修改脚本:$MS_PATH/RunCASTEP.sh -np 64 test1 test2 test3,test为导出文件test.param的文件名的前缀
2.Forcite和Mesocite模块的实现方式¶
1.Forcite和Mesocite时候,可以发现相应的路径下没有运行文件,是通过调用perl语言脚本的方式实现的,perl脚本的文件名与.xsd文件前缀一致,如创建名称test.pl,
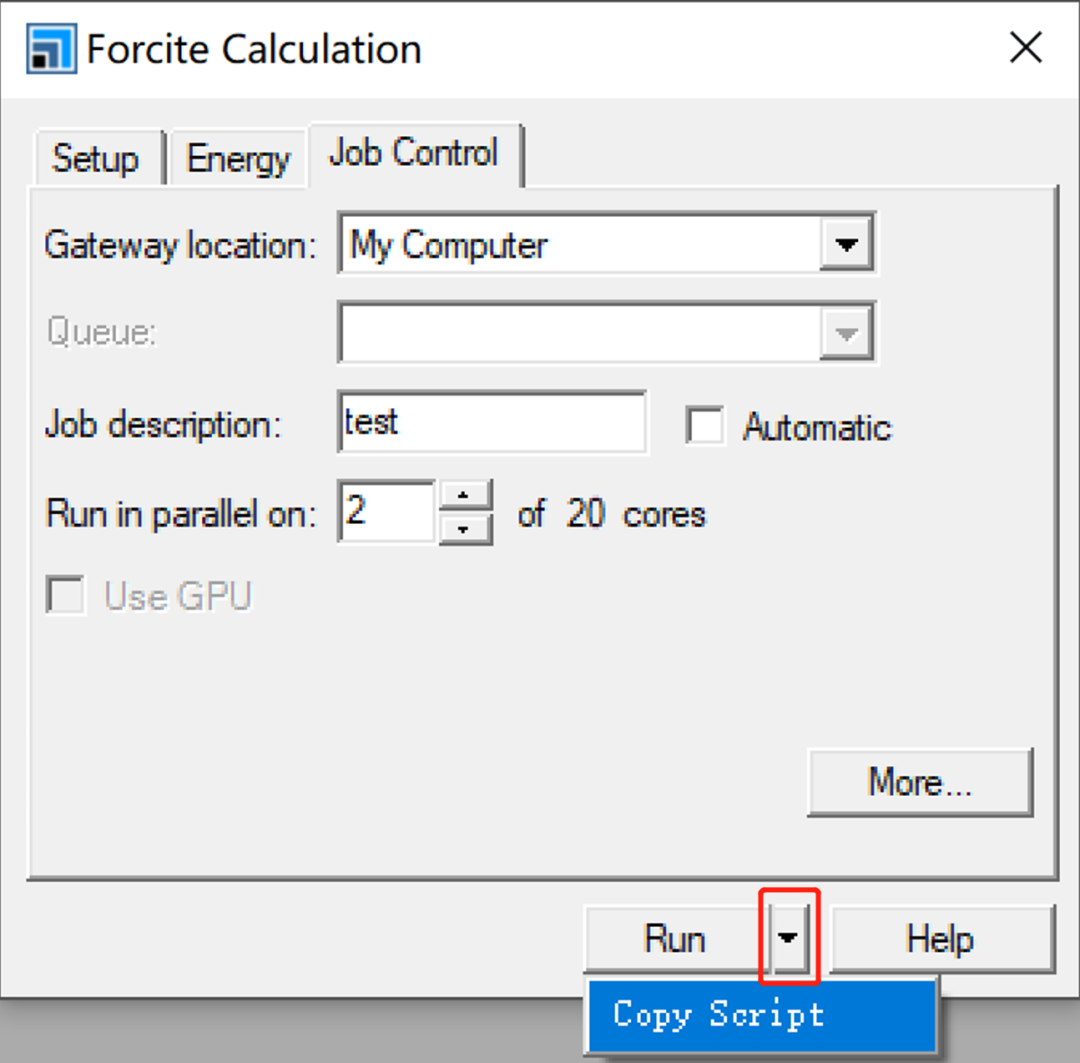
在MS的结构储存区域,新建脚本文件,将复制的脚本粘贴进去,但是这个脚本还不能进行计算,主要缺少计算对象的定义,所以需要在脚本中添加一行my $doc = $Documents{"test.xsd"};其中test为结构文件的名称;导出xsd文件和pl文件上传到作业目录
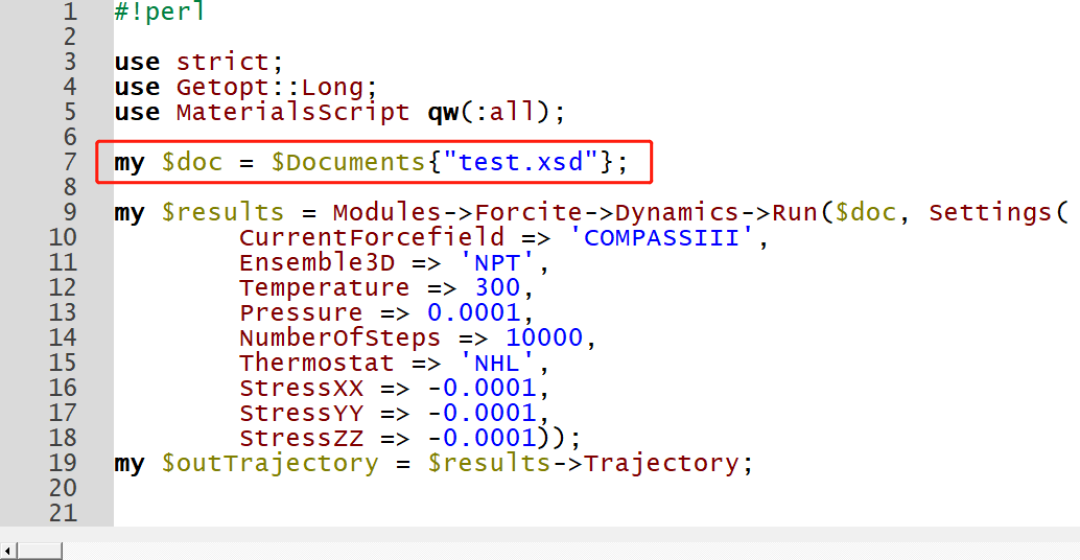
2.创建作业脚本ms.slurm,传输文件目录保持一致
#!/bin/bash
#SBATCH --job-name=test
#SBATCH --mem-per-cpu=8G
#SBATCH --partition=cpuXeon6458
#SBATCH --nodes=1
#SBATCH -n 64
#SBATCH --output=slurm_out.%j
#SBATCH --error=slurm_error.%j
module swap openmpi4 mvapich2/2.3.7
MS_PATH=/share/software/BIOVIA/MaterialsStudio23.1/etc/Scripting/bin
$MS_PATH/RunMatScript.sh -np 64 test > output #test为导出文件test.perl的文件名
使用sbatch ms.slurm提交作业
web平台提交¶
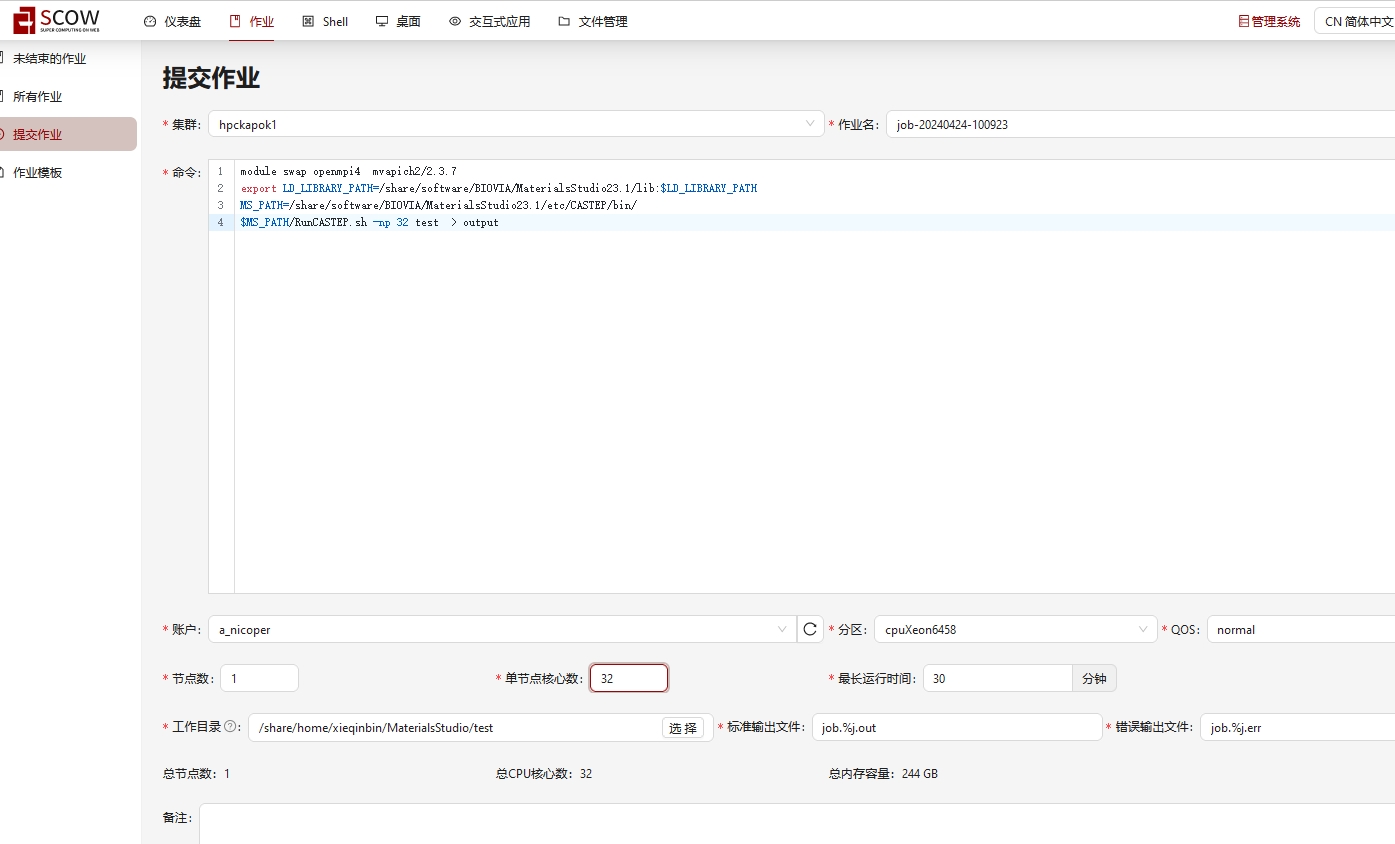
1.在“提交作业”页面填写作业信息 ,注意工作目录保持和脚本位置一致,申请核心数与命令中使用核心数-np保持一致
命令行解释¶
# 激活环境
module swap openmpi4 mvapich2/2.3.7
# 使用脚本计算
$MS_PATH/RunCASTEP.sh -np 32 test > output
# RunCASTEP.sh针对CASTEP模块,可根据/share/software/BIOVIA/MaterialsStudio23.1/etc路径选择其他模块
# test为windows上导出需要计算文件,如test.param,写为test即可
# 开始计算
sbatch <提交脚本>
论文致谢模板¶
(中文)本研究工作得到得到了华南理工大学科学计算公共服务平台的支持;
(英文)This work is partially supported by High Performance Computing Platform of South China University of Technology.
Contributor:qbxie