AlphaFold2¶
简介¶
AlphaFold2是Google旗下DeepMind开发的一款蛋白质结构预测程式。 该程序被设计为一个深度学习系统。 氨基酸折叠形成蛋白质。 AlphaFold人工智能有2个主要版本:AlphaFold 1(2018)和AlphaFold 2(2020)。
安装环境¶
序号 |
集群 |
平台 |
版本 |
位置 |
安装方式 |
|---|---|---|---|---|---|
1 |
hpckapok1 |
gpu |
AlphaFold2 |
/share/software/AlphaFold2/ |
conda |
2 |
hpckapok2 |
gpu |
AlphaFold2 |
/public/software/AlphaFold2/ |
容器镜像 |
使用方法¶
小技巧
命令运行:启动命令解释,参数之间使用空格分隔
slurm作业提交¶
hpckapok1¶
1.新建一个用于跑alphafold2的目录,复制alphafold-2.3.1到当前文件夹
mkdir alphafold2 && cd alphafold2
cp -r /share/software/Alphafold2/alphafold-2.3.1 ./
2.创建input文件夹,将输入文件test.fasta放置input文件夹下,示例:
>2LHC_1|Chain A|Ga98|artificial gene (32630)
PIAQIHILEGRSDEQKETLIREVSEAISRSLDAPLTSVRVIITEMAKGHFGIGGELASK
3.alphafold2文件夹下创建用于提交平台作业的脚本文件slurm.sh,以下为申请一张gpu卡进行计算
#!/bin/bash
#SBATCH --job-name=alphafold2
#SBATCH --partition=gpuA800
#SBATCH -N 1
#SBATCH -n 9
#SBATCH --gres=gpu:1
#SBATCH --output=%j.out
#SBATCH --error=%j.err
module load anaconda/3-2023.09
source activate af2
module load cuda/11.8
cd alphafold-2.3.1
./run_alphafold.sh -d /share/software/Alphafold2/alphafold_lib/data_tar -o ../output -f ../input/test.fasta -t 2023-03-12 -g true -c reduced_dbs
使用sbatch slurm.sh提交作业
4.作业结束后,输出结果在alphafold2/output/目录下
slurm作业提交¶
hpckapok2¶
1.编写alphafold2.slurm脚本
#!/bin/bash
#SBATCH -J STDIN_0112_164330
#SBATCH -p gpuA800
#SBATCH -N 1
#SBATCH --ntasks-per-node=1
#SBATCH --time 03:00:00
#SBATCH --comment=BASE
#SBATCH --array=1-1### MARK_MULTI_SUB
module load apps/anaconda3/3-2023.09
source activate af2
module load apps/singularity/3.9.9
python /public/software/AlphaFold2/alphafold-git/docker/run_alphafold232.py \
--fasta_paths <测试文件路径> \
--output_dir <输出结果路径> \
--db_preset=reduced_dbs \
--model_preset=multimer
2.使用sbatch alphafold2.slurm提交作业
web平台提交(salloc)¶
1.登录SCOW平台,并创建桌面

2.在桌面中打开终端

3.在终端申请计算节点资源,申请资源命令如下
# 申请资源命令例子,申请一个节点、cpuXeon6458资源分区、使用时长60分钟、32核
$ salloc -N 1 -p gpuA800 -t 60:00 --cpus-per-task=8 --gres=gpu:1
命令行参数解释:
-N <节点数量>
--cpus-per-task=<单进程 CPU 核心数>
--gres=gpu:<单节点 GPU 卡数>
-t <最长运行时间>
-p <使用的分区>
--qos=<使用的 QoS>
4.根据分配的计算节点,使用 ssh -Y <计算节点> 登录至计算节点。例如下图,分配的节点为comput1,则命令为: ssh -Y comput1

5.在打开的终端中运行以下命令,即可进行计算
module load apps/anaconda3/3-2023.09
source activate af2
module load apps/singularity/3.9.9
python /public/software/AlphaFold2/alphafold-git/docker/run_alphafold232.py \
--fasta_paths <测试文件路径> \
--output_dir <输出结果路径> \
--db_preset=reduced_dbs \
--model_preset=multimer
6.新建一个终端并登录至计算节点,使用 nvidia-smi 命令即可查看GPU显卡使用状态
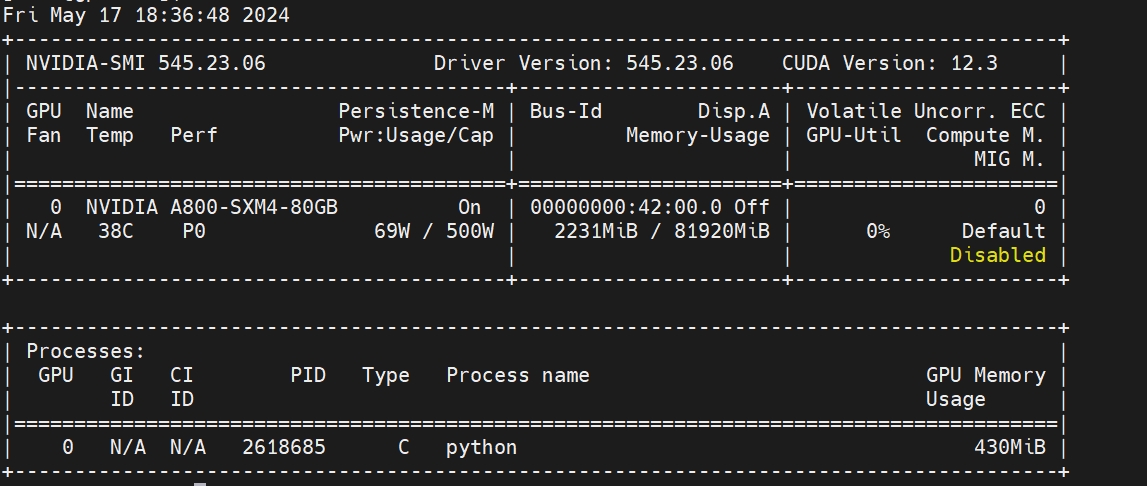
测试样例¶
测试文件路径:/public/software/share/case/alphafold/test.fasta
测试命令:
# 激活环境
module load apps/anaconda3/3-2023.09
source activate af2
module load apps/singularity/3.9.9
# 创建结果保存路径
mkdir -vp $HOME/alp-test/res
# 启动计算
python /public/software/AlphaFold2/alphafold-git/docker/run_alphafold232.py \
--fasta_paths /public/software/share/case/alphafold/test.fasta \
--output_dir $HOME/alp-test/res-1 \
--db_preset=reduced_dbs \
--model_preset=multimer
Contributor:B君、qbxie